DNA甲基化 (5-甲基胞嘧啶) 和RNA甲基化 (N6-甲基腺嘌呤) 是两种重要的表观遗传学调控,在调控基因表达、细胞的分化与生长发育等过程中发挥着关键作用。N6-甲基腺嘌呤参与调控林木干旱胁迫 (Gao et al., 2022) 和次生木质部发育 (Gao et al., 2021), 林木中过表达甲基转移酶MTA可以产生更发达的根系并增强干旱耐受性 (Lu et al., 2020)。水稻中过表达RNA去甲基化酶使得产量与生物量增加了50% (Yu et al., 2021)。快速生长是毛竹笋较为重要的生物学特性之一,毛竹茎秆的快速生长得益于每个节间细胞的快速分裂和伸长 (Chen et al., 2022)。因此系统的研究毛竹快速生长期间单节间的甲基化修饰动态变化将有助于阐明毛竹笋快速生长的表观遗传调控机制, 针对毛竹笋快速生长的甲基化调控进行深入研究可为禾本科作物遗传改良提供借鉴意义。
近日,福建农林大学林学中心顾连峰教授研究组在Plant Physiology发表了题为“Comprehensive profiling of epigenetic modifications in fast-growing moso bamboo shoots”的研究论文,对不同高度(2米和4米)毛竹笋单节间(基部和中部)进行了甲基化调控生长相关研究。RNA甲基化研究揭示2米毛竹笋具有更高的N6-甲基腺嘌呤修饰比例, DNA甲基化研究揭示4米毛竹笋具有更高5-甲基胞嘧啶水平,具有更短Poly(A)尾长度的基因其CG类型的DNA甲基化水平也更低。

本研究通过石蜡切片结合多组学手段对快速生长过程中不同高度毛竹笋单节间进行了全面研究,发现竹笋生长过程中具有如下动态变化规律:(1) 2米毛竹笋第十八节间的细胞排列紧凑,能观察到大量细胞核,而4米毛竹笋第十八节间的细胞明显纵向伸长,细胞核出现概率显著降低。(2) 随着毛竹笋高度的增加,木质素调控通路相关基因和糖基转移酶43家族基因表达量大幅上升,从而导致毛竹笋的快速生长过程伴随着笋的高度木质化和纤维化。(3) 毛竹笋快速生长过程中伴随着复杂的转录后调控。选择性多聚腺苷酸化分析结果显示随着快速生长的进行毛竹笋基部的3′ UTR长度显著缩短。(4) 毛竹笋快速生长期间Poly(A)尾长度呈现动态变化,2米毛竹笋具有更长的Poly(A)尾巴长度,Poly(A)尾巴长度显著变化的基因主要与蛋白质翻译和囊泡融合有关。此外,关联分析显示发生内含子保留和外显子跳跃的读段具有更长的Poly(A)尾。(5) N6-甲基腺嘌呤分析结果显示2米毛竹笋具有更高的m6A修饰比例。组成型m6A修饰位点和特异性m6A修饰位点展示出了不同的分布趋势,组成型修饰位点更多富集于3′ UTR附近,特异性修饰位点则更多富集于CDS区域。组成型修饰位点所在的基因大多为与细胞质翻译和核糖体组装相关的管家基因,而特异性修饰位点所在的基因则随着快速生长阶段的不同发挥不同的功能。此外,木质素调控通路相关基因的m6A修饰率都低于该时期的平均水平。(6) 全基因组重亚硫酸盐测序结果显示4米毛竹笋具有更高的DNA甲基化水平。转座元件在整个快速生长期间均维持了较高的甲基化水平。差异甲基化(DMR)分析结果显示含有CG-DMRs的编码基因最多,大部分DMRs所在的基因主要与微管运动、RNA合成与降解有关。此外,关联分析显示Poly(A)尾长度与编码基因区间内的CG甲基化水平呈正相关。
本研究对快速生长过程中毛竹笋单节间不同部位的甲基化修饰和转录后调控机制进行了系统分析,并对这些表观遗传途径的内在联系进行了初步探索 (图一)。为今后借助于遗传转化的手段探究禾本科植物生长发育的表观调控机制提供了前期基础。
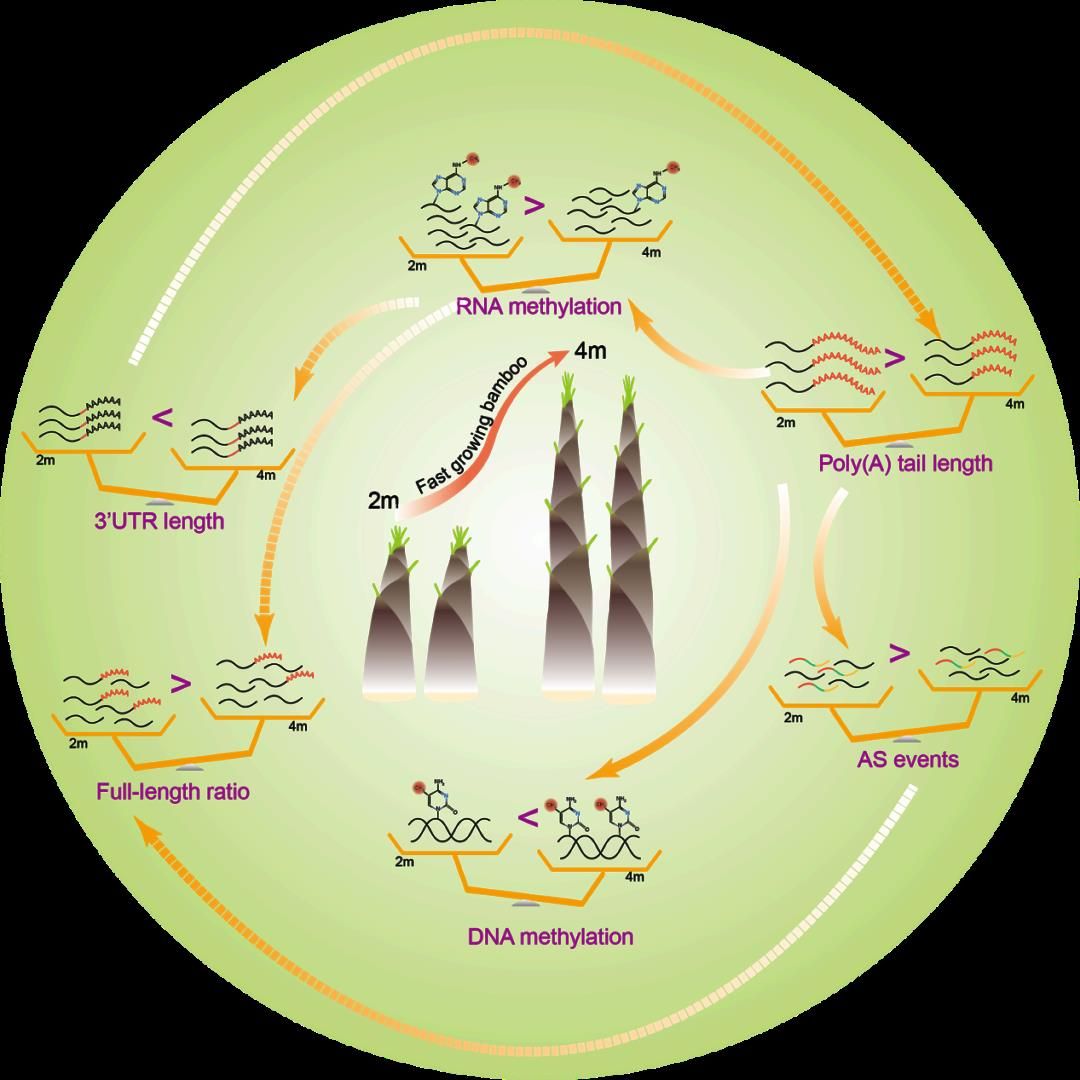
图一 毛竹笋快速生长过程中的表观遗传学调控模式图
福建农林大学林学院研究生李涛、王慧慧和张娅欣为共同第一作者,林学院王汇源、张泽宇和刘旭庆等博士生也参与了本工作,林学中心顾连峰教授为通讯作者。该研究得到了十四五国家重点研发项目(2021YFD2200505)、基金委面上项目(31971734)和福建农林大学林学院高峰学科的支持(72202200205)。
相关文献:
Chen, M., Guo, L., Ramakrishnan, M., Fei, Z., Vinod, K.K., Ding, Y., Jiao, C., Gao, Z., Zha, R., Wang, C., Gao, Z., Yu, F., Ren, G. and Wei, Q. (2022) Rapid growth of Moso bamboo (Phyllostachys edulis): Cellular roadmaps, transcriptome dynamics, and environmental factors. The Plant Cell, 34, 3577-3610.
Gao, Y., Liu, X., Jin, Y., Wu, J., Li, S., Li, Y., Chen, B., Zhang, Y., Wei, L., Li, W., Li, R., Lin, C., Reddy, A.S.N., Jaiswal, P. and Lianfeng, G. (2022) Drought induces epitranscriptome and proteome changes in stem-differentiating xylem of Populus trichocarpa. Plant Physiology, 190, 459-479.
Gao, Y., Liu, X., Wu, B., Wang, H., Xi, F., Kohnen, M.V., Reddy, A.S. and Gu, L. (2021) Quantitative profiling of N6-methyladenosine at single-base resolution in stem-differentiating xylem of Populus trichocarpa using Nanopore direct RNA sequencing. Genome Biology, 22, 1-17.
Lu, L., Zhang, Y., He, Q., Qi, Z., Zhang, G., Xu, W., Yi, T., Wu, G. and Li, R. (2020) MTA, an RNA m6A methyltransferase, enhances drought tolerance by regulating the development of trichomes and roots in poplar. International journal of molecular sciences, 21, 2462.
Yu, Q., Liu, S., Yu, L., Xiao, Y., Zhang, S., Wang, X., Xu, Y., Yu, H., Li, Y. and Yang, J. (2021) RNA demethylation increases the yield and biomass of rice and potato plants in field trials. Nature Biotechnology, 39, 1581-1588.
论文链接: https://doi.org/10.1093/plphys/kiac525











