N6 -甲基腺苷(m6A)作为一种动态可逆的转录后标记物,在多种生命活动的调控中起着至关重要的作用。m6A的大部分生物学功能是由m6A调控元件介导的,包括m6A甲基转移酶(writers)、去甲基酶(erasers)和阅读蛋白(readers)【1】。m6A甲基转移酶催化mRNA上的腺苷酸发生m6A修饰,而去甲基酶对已发生m6A修饰的碱基进行去甲基化修饰,从而保证细胞内m6A修饰水平的动态平衡【1】。m6A结合蛋白与m6A修饰的RNA特异结合,从而实现甲基化修饰的生物学功能。writers、erasers和readers构成了一个复杂的调控系统,指导m6A的形成、删除和解码【1-3】。
m6A修饰的写入是由一系列m6A甲基转移酶组件形成的蛋白质复合体控制的。在拟南芥中,m6A甲基转移酶组分包括MTA、MTB、FIP37、VIR和HAKAI。MT-A70家族是最早在植物中发现的m6A甲基转移酶组分,在高等真核生物中可分为3个亚家族: MTA(人类METTL3同源蛋白)、MTB(人类METTL14同源蛋白)和MTC(人类METTL4同源蛋白)。最早报道的m6A去甲基酶是动物中的FTO【2-3】和ALKBH5,它们是ALKBH家族的成员。ALKBH家族在拟南芥中有14个成员: ALKBH1A-D、ALKBH2、ALKBH6、ALKBH8A-B、ALKBH9A-C和ALKBH10A-C,分为7个主要分支。m6A阅读蛋白属于YTH家族成员。YTH家族成员高度保守,包含一个具有芳香笼的YTH结构域,用于m6A识别。在人类中,YTH家族有5个成员,而在植物中,YTH家族成员的数量显著增加。
近日,福建农林大学林学中心顾连峰教授课题组在The Plant Journal期刊在线发表了题为Evolution and post-transcriptional regulation insights of m6A writers, erasers and readers in plant epitranscriptome的论文,利用系统进化研究和纳米孔单分子测序技术,对植物中m6A调控元件及其修饰进行了全面的进化和功能分析。

该研究首先从13 种绿藻、7 种轮藻、7 种苔藓、1 种石松、3 种蕨类、9 种裸子和114 种被子植物中鉴定到了4062 个m6A调控元件,包含512 个MT-A70,200 个FIP37,145 个VIR,148 个HAKAI,1615 个ALKBH,1496 个 YTH 蛋白 (图一)。其中m6A甲基转移酶的各个组分(包括MT-A70、FIP37、VIR 和 HAKAI)的数目都比较保守,ALKBH家族的数目从低等到高等植物中存在着轻微的扩张现象, 而YTH家族存在着明显的扩张现象。
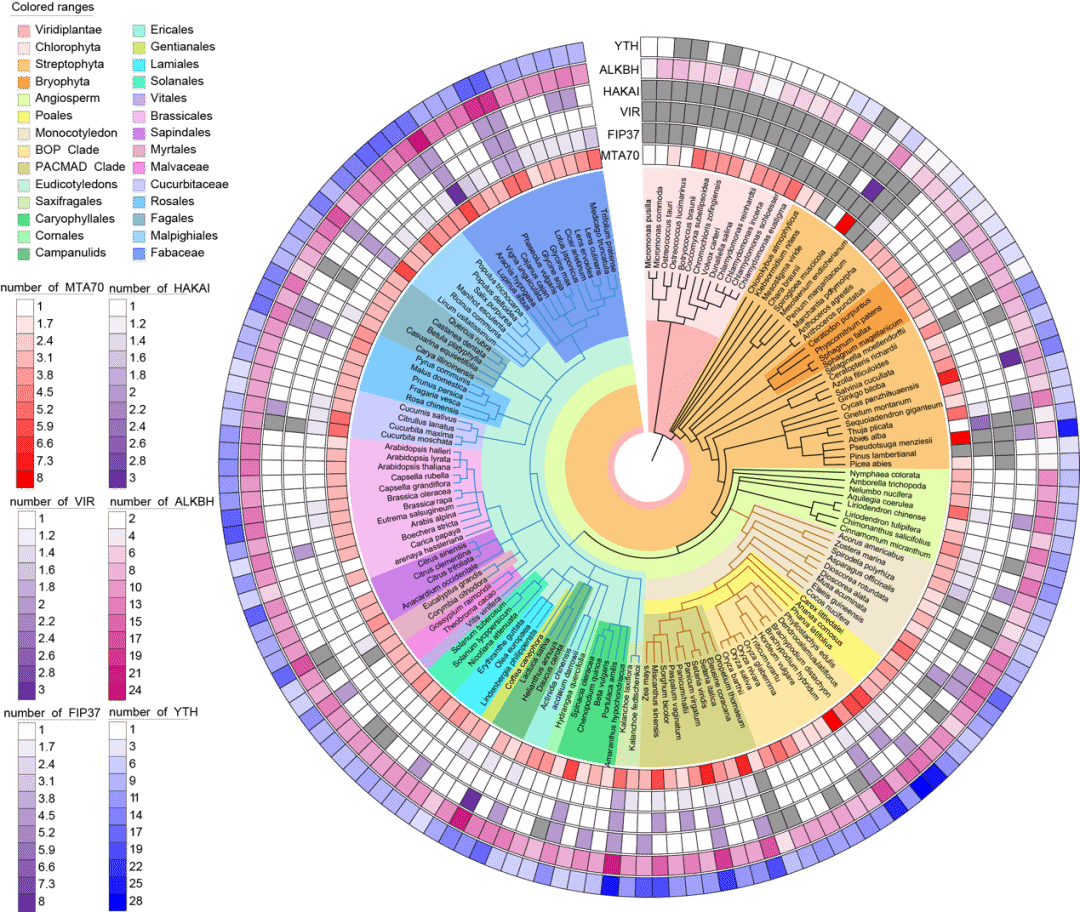
图一: 154 个物种中m6A甲基转移酶、去甲基酶和结合蛋白的数目
该研究将所有鉴定到的m6A调控元件划分成了两个数据集分别进行系统进化分析,其中数据集1 包括了所有低等的植物和4 种代表性的被子植物,数据集2 包括了所有的被子植物。对系统进化的结果进行推断,得到了三大类m6A调控元件的系统进化模型(图二):(1)MT-A70家族:在藻类植物和陆地植物的共同祖先中分别演化出 MTA,MTB,MTC,Chlorophytes-1 和Chlorophytes-2 五个分支。Chlorophytes-1 和Chlorophytes-2 在陆地植物和轮藻中丢失。MTB分支在禾本科中发生了扩张,产生了MTB-Poaceae-1 和MTB-Poaceae-2 两个亚支;(2)ALKBH家族:在藻类植物和陆地植物的共同祖先中分别演化出八个分支:ALKBH1A,ALKBH1B/1C1D,ALKBH2,ALKBH6,ALKBH8A,ALKBH8B,ALKBH9A/9B/9C/10A/10B/10C和algae-specific。algae-specific分支在陆地植物中丢失。随后,在轮藻分化之后,裸子植物分化之前,ALKBH9A/9B/9C和ALKBH10A/10B/10C发生分化。被子植物最终扩展成九个主要的分支。ALKBH1B/1D、ALKBH9B、ALKBH10B、ALKBH10C 在某些被子植物中拷贝数也有增加;(3)YTH家族:早在藻类植物和陆地植物的共同祖先中,YTH 家族就演化出了 YTHDF 和 YTHDC 两支,随后,在绿藻分化之后,轮藻分化之前,YTHDC 演化成 CPSF30 和 ECT12 两支。在轮藻分化之后,YTHDF 发生分化,并在被子植物中最终扩展成了 ECT1/2、ECT5/9/10、ECT6、ECT8、ECT11 五个分支。最终,被子植物中形成了七个主要的分支。在 ECT1/2 和 ECT5/9/10 分支中,双子叶和单子叶植物中均发生了较大的扩张,ECT1/2 最终形成了2 个双子叶亚支和4 个禾本科亚支,ECT5/9/10 最终形成了4 个双子叶亚支和3 个禾本科亚支。在ECT6 分支中,单子叶植物也发生扩张,形成了3 个禾本科亚支。除此之外,这些亚支中还在不同进化分类群上存在许多谱系特异性的基因重复和丢失事件。值得注意的是,ECT12 在单子叶植物植物中发生了丢失。

图二: m6A调控元件的系统发育分析。(A)系统发育分析流程示意图;(B)使用来自4 种被子植物和40 种非被子植物的133 个MT-A70蛋白构建的绿色植物中MT-A70家族的系统发育树;(C)使用来自114 种被子植物的393 个MT-A70蛋白构建的被子植物中MT-A70家族的系统发育树;(D) 使用来自4 种被子植物和40 种非被子植物的212 个ALKBH蛋白构建的绿色植物中ALKBH家族的系统发育树;(E) 使用来自114 种被子植物的1294 个ALKBH蛋白构建的被子植物中ALKBH家族的系统发育树;(F) 使用来自4 种被子植物和40 种非被子植物的227 个YTH蛋白构建的绿色植物中YTH家族的系统发育树;(G) 使用来自114 种被子植物的1315 个YTH蛋白构建的被子植物中YTH家族的系统发育树。
保守的基因组上下文信息为比较进化分析提供了关键信息,由于非被子植物与被子植物进化距离上较远,为了深入研究被子植物的共线性结果,该研究进一步使用114 种被子植物对MTA70,FIP37,VIR,HAKAI,ALKBH 和 YTH进行共线性网络的研究,揭示了被子植物m6A调控元件的保守基因组上下文 (图三)。
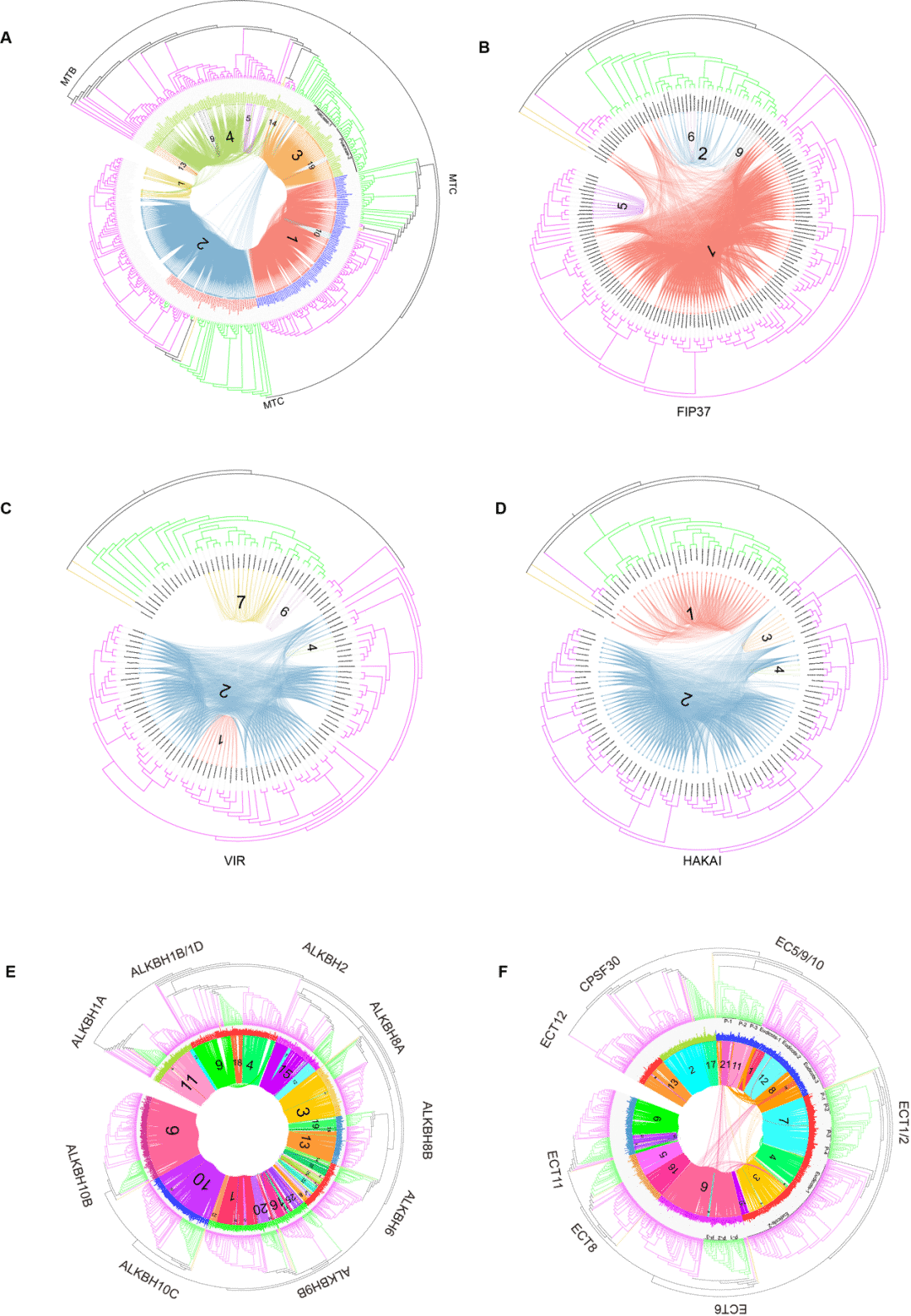
图三: 被子植物中(A)MT-A70s,(B)FIP37s,(C)VIRs,(D)HAKAIs,(E)ALKBHs, (F)YTHs的系统进化和共线性关系除此之外,研究人员还利用纳米孔单分子测序数据绘制了六个被子植物的poly(A)尾巴长度和m6A修饰图谱。发现基因表达和poly(A)尾巴长度负相关,与m6A修饰负相关 (图四)。同时poly(A)尾巴长度和m6A修饰呈正相关。并且该趋势在六个物种中均一致,暗示基因表达、poly(A)尾巴长度和m6A修饰的相互关联在被子植物中是保守的。

图四: 六种被子植物的poly(A)尾巴长度和m6A修饰图谱。(A)不同物种中整体的读段poly(A)尾巴长度、基因poly(A)尾巴长度中位数、修饰位点的m6A 修饰率和基因的m6A修饰率中位数分布;(B)散点图展示了基因表达和poly(A)尾巴长度的关联;(C)散点图展示了基因表达和m6A 修饰的关联;(D)箱形图展示了有m6A修饰和没有m6A修饰的基因的poly(A)尾巴长度;(E)折线图展示了基于基因poly(A)尾巴长度排序的m6A修饰率分布;热图展示了不同物种直系同源基因之间的(F)基因表达水平,(G)poly(A)尾巴长度和(H)m6A修饰皮尔逊相关性。
进一步对三大类调控基因的研究发现,同一进化分支的基因往往有着相似的转录及转录后调控模式。特别是,观察到共线性网络研究中发现的与其他亚支连接松散的ECT1/2-Poaceae4与其他亚支相比,明显高度表达,并且修饰率明显降低,poly(A)尾巴长度轻微缩短 (图五)。在ECT1/2-Poaceae4亚支的基因的 3’UTR区域中观察到了保守的修饰位点。将这些修饰位点进行A到G的突变后,荧光值明显强于未突变的区域。这提示3’UTR区域中保守的修饰位点可以调控 LUC的表达。
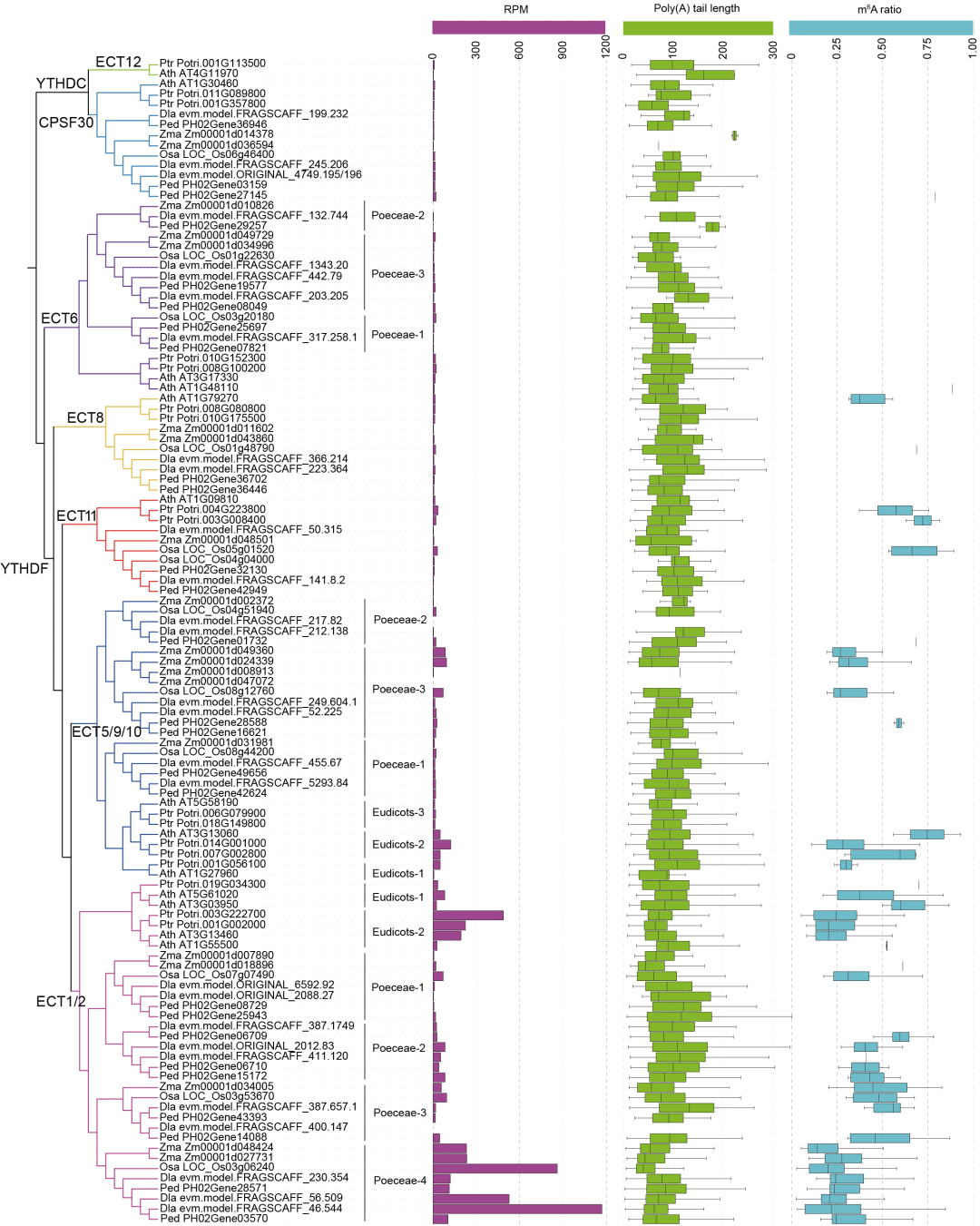
图五: 拟南芥、杨树、玉米、水稻、毛竹和麻竹中m6A阅读蛋白的转录及转录后调控分析。从左至右分别为六种被子植物YTH的系统进化树、基因表达直方图、Poly(A)尾巴长度箱线图和m6A修饰率箱线图。最后,该研究在五个物种中构建了调控m6A调控元件的转录因子网络。例如拟南芥BPC1及其在其他物种中的同源基因作都作用于YTH家族中ECT1/2分支的成员 (图六)。进一步利用酵母单杂证实了BPC1和PheECT1/2-5之间的互作关系。
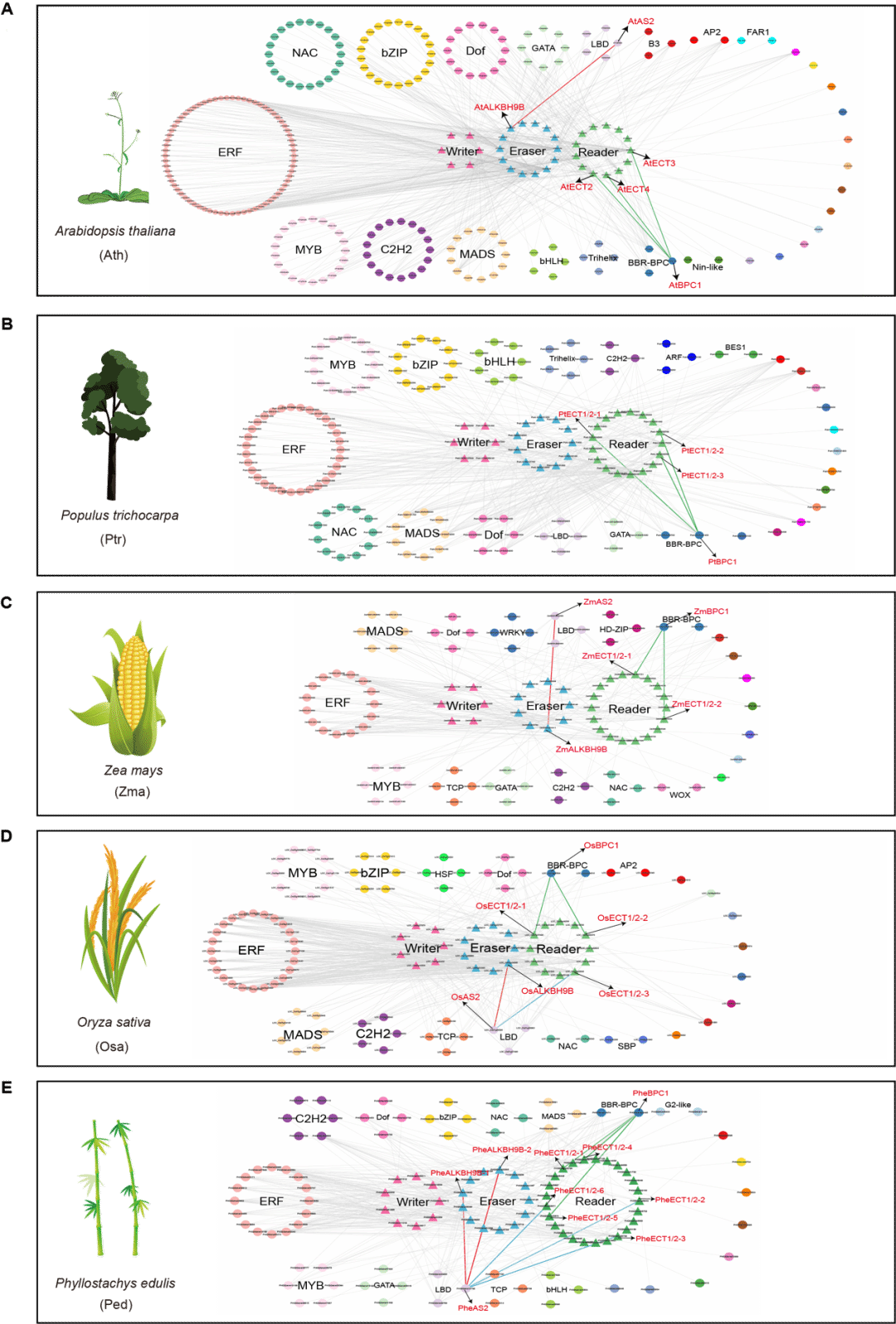
图六: 拟南芥、杨树、玉米、水稻和毛竹中m6A调控元件的转录因子网络
总之,该研究鉴定了154 种植物中4062 个m6A调控元件,利用大规模系统发育方法探索其进化,并提出了植物中MTA-70、ALKBH和YTH推断的进化模型 (图七)。随后,利用共线性网络分析进一步探索了114 种被子植物中m6A调控元件的保守基因组上下文。此外,利用纳米孔单分子测序技术揭示了6 种被子植物物种的Poly(A)尾巴长度和m6A修饰分布,并特别关注了m6A调控元件的分布(图七)。这为将来进一步探究m6A的调控提供了参考。
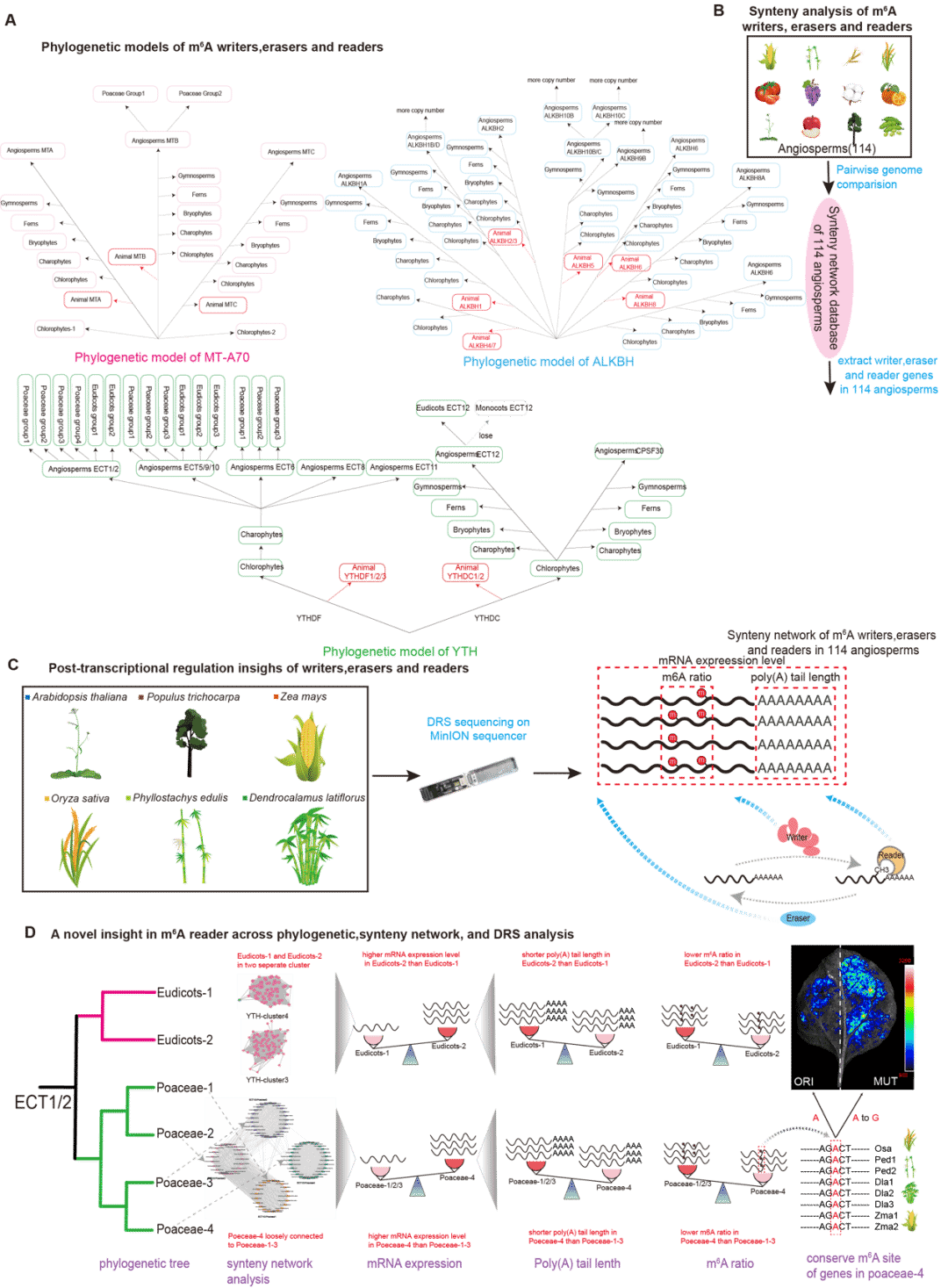
图七 本研究的简要总结。(A)推断的MT-A70、ALKBH和YTH家族在植物中的进化模型;(B)共线性网络分析。共线性网络分析表明,ECT1/2-Eudicots1和ECT1/2-Eudicots2被分别聚成了两个集群,ECT1/2-Poaceae4与ECT1/2-Poaceae1-3分支连接松散。系统进化分类和共线性网络模式的差异可能是导致ECT1/2-Eudicots2和ECT1/2-Poaceae4分支中基因表达升高的原因;(C)m6A甲基转移酶、去甲基酶和阅读蛋白的转录后调控见解。与其他分支相比,ECT1/2-Eudicots2和ECT1/2-Poaceae4分支的m6A修饰明显较低,基因的Poly(A)尾巴长度略微缩短,这可能是造成这些分支中基因表达的升高的另一个原因;(D)基于系统发育、共线性网络和DRS数据的综合分析的新见解。
福建农林大学未来学院硕士研究生张珺、林学院博士生吴林和林学院本科生慕乐乐为论文共同第一作者,林学中心顾连峰教授为论文通讯作者。该研究得到十四五国家重点研发项目、基金委面上项目和福建农林大学林学院高峰学科的支持。
参考文献:1. Yue H, Nie X, Yan Z, et al. N6-methyladenosine regulatory machinery in plants: composition, function and evolution[J]. Plant Biotechnol J, 2019, 17(7): 1194-1208.2. Jia G, Fu Y, Zhao X, et al. N6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO[J]. Nat Chem Biol, 2011, 7(12): 885-887.3. Yu Q, Liu S, Yu L, et al. RNA demethylation increases the yield and biomass of rice and potato plants in field trials[J]. Nat Biotechnol, 2021, 39(12): 1581-1588.4. Jia J, Lu W, Liu B, et al. An atlas of plant full-length RNA reveals tissue-specific and monocots-dicots conserved regulation of poly(A) tail length[J]. Nat Plants, 2022, 8(9): 1118-1126.5. Miao Z, Zhang T, Xie B, Qi Y, Ma C. Evolutionary Implications of the RNA N6-Methyladenosine Methylome in Plants. Mol Biol Evol. 2022 Jan 7;39(1):msab299.6. Parker M T, Knop K, Sherwood A V, et al. Nanopore direct RNA sequencing maps the complexity of Arabidopsis mRNA processing and m6A modification[J]. Elife, 2020, 9:e49658.7.Gao Y, Liu X, Wu B, et al. Quantitative profiling of N6-methyladenosine at single-base resolution in stem-differentiating xylem of Populus trichocarpa using Nanopore direct RNA sequencing[J]. Genome Biol, 2021, 22(1): 22.8. Zhao T, Holmer R, De Bruijn S, et al. Phylogenomic synteny network analysis of MADS-Box transcription factor genes reveals lineage-specific transpositions, ancient tandem duplications, and deep positional conservation[J]. Plant Cell, 2017, 29(6): 1278-1292.
全文链接:https://doi.org/10.1111/tpj.16996











